博文
用R分析COVID-19流行病学[译文]
||
作者简介:Tim Churches是新南威尔士大学临床医学院的高级研究员,也是Ingham应用医学研究所的健康数据科学家。 他的研究领域涉及普通医学、全科医学、职业卫生和公共卫生实践,尤其是人口健康监测和临床流行病学。
英文原文的链接:COVID-19 epidemiology with R
前言
当我在2020年3月4日撰写本文时,世界正处于由SARS-Cov2病毒引起的全球COVID-19大流行的风口浪尖。 每个新闻报道都以令人震惊且不断增长的全球累计病例数和因COVID-19造成的死亡人数为主导。 遍布全球的仪表盘像圣诞树一样开始亮起来。
对于R用户来说,浅显的问题:“ R可以提供帮助了解情况的信息吗?”
答案是:“是的,很多!”
实际上,R是暴发流行病学家选择的工具之一,可在CRAN和其他地方快速搜索到许多专门用于暴发管理和分析的R程序包。 这篇文章并不是要评论可用的软件包,而是要展示R Epidemics联盟(RECON)站点上一些优秀程序包的实用性,以及将使用R和tidyverse包进行数据采集、整理和可视化。这篇文章基于我在过去几周发表的两篇关于同一主题的更长更详细的博客文章,但使用了美国数据。
数据采集
获得的COVID-19流行病的详细、准确和最新的数据并不像看上去那么简单。 受影响的各个国家及其省/政府网站都提供了因病毒引起的病例数、治愈数和死亡数的详实汇总数据,但这些数据往往是嵌入(通常为非英语)文本的计数形式。
从这些政府网站上提取和整理的数据有几个潜在来源。一个广泛使用的数据源是由约翰霍普金斯大学系统科学与工程中心(JHU CCSE)整理的数据集,它用作上述仪表板的数据源。它非常容易使用:只需从相应的GitHub URL读取CSV文件即可。然而,它缺乏细节(这不是它的预期目的),当作为不同的每日病例时间序列检查时,它含有相当多的缺失或异常数据点,这是一个相对较小的问题,在这里将进一步探讨。
另一套便捷的资源是相关的维基百科页面,例如针对中国的维基百科页面。日本、韩国、伊朗、意大利和许多其他国家/地区都有相应的页面。这些Wikipedia页面往往更加详细,并且可以很好地索引回原始的源网页,但是对于Web抓取来说,它们具有很大的挑战性,因为随着不同wikipedia贡献者调整它们的外观,出现数据的表格格式经常改变。尽管如此,我们还是会从合适的维基百科页面上抓取到有关美国(截至3月4日)已确诊的COVID-19病例的详细数据。另一个好处是维基百科页面是版本化的,因此可以从特定版本的表中收集数据。 但是,如果您想每天更新数据,则使用Wikipedia页面作为源进行Web抓取代码,并且每天维护。
约翰霍普金斯大学整理的病例数据
获取这些数据很容易。他们提供的时间序列格式对我们而言是最方便的。我们将删除与“钻石公主”号游轮相关的美国病例,因为我们可以假定这些病例在遣返时被隔离在家中,因此不太可能引发更多病例。我们还将JHU数据中的日期前移一天,以近似反映美国时区,因为原始日期是相当于午夜的格林威治时间。这是必要的,因为我们将把JHU数据与源自维基百科的美国数据结合在一起,这些数据是根据参考美国当地时区的日期制成的表格。
我们还需要区分JHU数据(以累积计数形式提供),以获取每日病例数。每日病例数在流行病学方面比累积计数更有用。dplyr包将轻松处理这些工作。
jhu_url <- paste("https://raw.githubusercontent.com/CSSEGISandData/",
"COVID-19/master/csse_covid_19_data/", "csse_covid_19_time_series/",
"time_series_19-covid-Confirmed.csv", sep = "")
us_confirmed_long_jhu <- read_csv(jhu_url) %>% rename(province = "Province/State",
country_region = "Country/Region") %>% pivot_longer(-c(province,
country_region, Lat, Long), names_to = "Date", values_to = "cumulative_cases") %>%
# adjust JHU dates back one day to reflect US time, more or less
mutate(Date = mdy(Date) - days(1)) %>% filter(country_region ==
"US") %>% arrange(province, Date) %>% group_by(province) %>%
mutate(incident_cases = c(0, diff(cumulative_cases))) %>%
ungroup() %>% select(-c(country_region, Lat, Long, cumulative_cases)) %>%
filter(str_detect(province, "Diamond Princess", negate = TRUE))
现在可以使用ggplot2可视化JHU数据,来汇总全美疫情: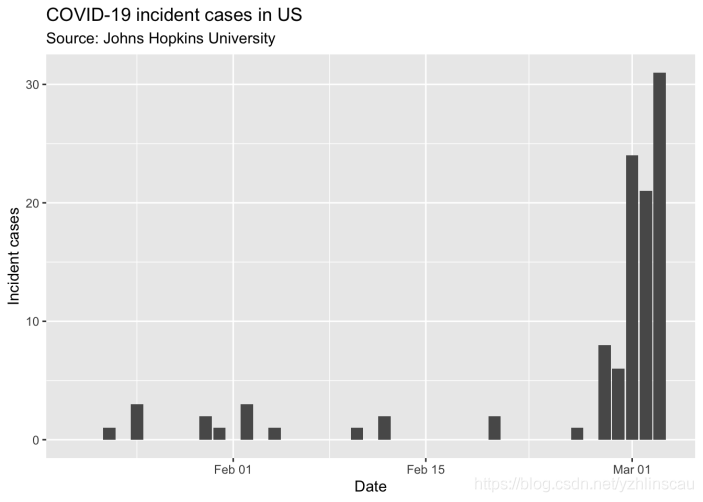
因此,目前尚无大量数据可使用。关于这些病例是否是输入性的信息是缺失的,也就是说,感染很可能是在美国境外获得的,或者是由于本地传播(可能来自输入性病例)而导致的本地感染。
抓取维基百科
Wikipedia上的美国COVID-19页面含有几个表格,其中一个表格列出了美国所有最初的病例的,撰写本文时,截至3月2日,这些表格似乎已经相当完整。我们将利用tidyverse包组件的出色rvest软件包从这个版本的Wikipedia页面中抓取此表。再次,我们需要进行一些数据整理,使得解析后的表变成可用的格式。
# the URL of the wikipedia page to use is in wp_page_url
wp_page_url
## [1] "https://en.wikipedia.org/w/index.php?title=2020_coronavirus_outbreak_in_the_United_States&oldid=944107102"# read the page using the rvest package.outbreak_webpage <- read_html(wp_page_url)# parse the web page and extract the data from the eighth table
us_cases <- outbreak_webpage %>% html_nodes("table") %>% .[[8]] %>%
html_table(fill = TRUE)
# The automatically assigned column names are OK except that
# instead of County/city and State columns we have two
# columns called Location, due to the unfortunate use of# colspans in the header row. The tidyverse abhors
# duplicated column names, so we have to fix those, and make
# some of the other colnames a bit more tidyverse-friendly.
us_cases_colnames <- colnames(us_cases)
us_cases_colnames[min(which(us_cases_colnames == "Location"))] <- "CityCounty"
us_cases_colnames[min(which(us_cases_colnames == "Location"))] <- "State"
us_cases_colnames <- us_cases_colnames %>% str_replace("Location",
"CityCounty") %>% str_replace("Location", "State") %>% str_replace("Case no.",
"CaseNo") %>% str_replace("Date announced", "Date") %>% str_replace("CDC origin type",
"OriginTypeCDC") %>% str_replace("Treatment facility", "TreatmentFacility")
colnames(us_cases) <- us_cases_colnames
# utility function to remove wikipedia references in square brackets
rm_refs <- function(x) stringr::str_split(x, "\\[", simplify = TRUE)[, 1]
# now remove references from CaseNo column, convert it to
# integer, convert the date column to date type and then lose
# all rows which then have NA in CaseNo or NA in the date column
us_cases <- us_cases %>% mutate(CaseNo = rm_refs(CaseNo)) %>%
mutate(CaseNo = as.integer(CaseNo), Date = as.Date(parse_date_time(Date,
c("%B %d, %Y", "%d %B, %Y")))) %>% filter(!is.na(CaseNo),
!is.na(Date)) %>%
# convert the various versions of unknown into NA in the OriginTypeCDC
columnmutate(OriginTypeCDC = if_else(OriginTypeCDC %in% c("Unknown",
"Undisclosed"), NA_character_, OriginTypeCDC))
至此,我们有了一个可用的净表,如下所示(略):
我们不用费心清理所有列,因为这里只使用其中的一些。
因此我们将使用2月20日之前的Wikipedia数据,那之后使用JHU计数,但是我们将使用2月20日之后的Wikipedia数据将JHU计数分为与旅行或多或少相关。这将为我们提供一个数据集,至少在数据源的完整程度上可以区分本地病例和输入病例。 暴发流行病学涉及足够的实践练习。
让我们可视化两个来源以及合并后的数据集: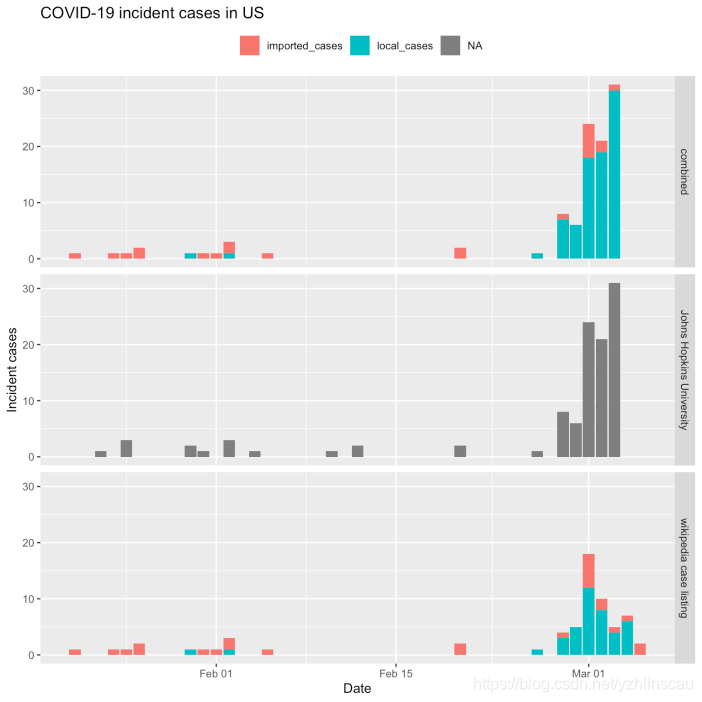
用EarlyR和EpiEstim包进行分析
顾名思义,earlyR软件包旨在疾病爆发早期用于计算几个关键统计量。特别是earlyR中的get_R()函数计算了再生数(reproduction number)的最大似然估计,即每个感染者引起的新病例的平均数。EpiEstim包中的total_infectivity()函数计算
λ值,它是当前“感染力”或爆发传染性的相对度量:
λ=∑t−1s=1ysw(t–s)
式中,w()是序列间隔的概率质量函数(PMF),ys是时间s的发生率。如果λ值正在下降,则是好兆头;如果不是,则是坏兆头。
这些计算的关键参数是连续间隔(SI)的分布,该间隔是指从病例出现症状的日期到引起该病例的任何继发病例的发病日期之间的时间。通常假定这些序列间隔是以平均值和标准差为参数化的离散γ分布,尽管更复杂的分布可能更现实。 关于序列间隔的更详细讨论,以及行列表数据的首要作用,请参阅先前文章。
目前,我们仅使用平均值为5.0天、标准差为3.4的离散γ分布。 该平均值低于中国疫情爆发早期公布的估计值,但似乎更接近于COVID-19病毒的行为(基于WHO召开COVID-19电话会议的知情人士的个人通讯) 。显然,应该使用不同但合理的序列间隔分布进行敏感性分析,但为简化起见,在此我们将其省略。
请注意,仅使用本地病例来计算λ值。如果我们仅使用JHU数据(包括本地病例和输入病例),则估计的
λ值将有向上的偏差。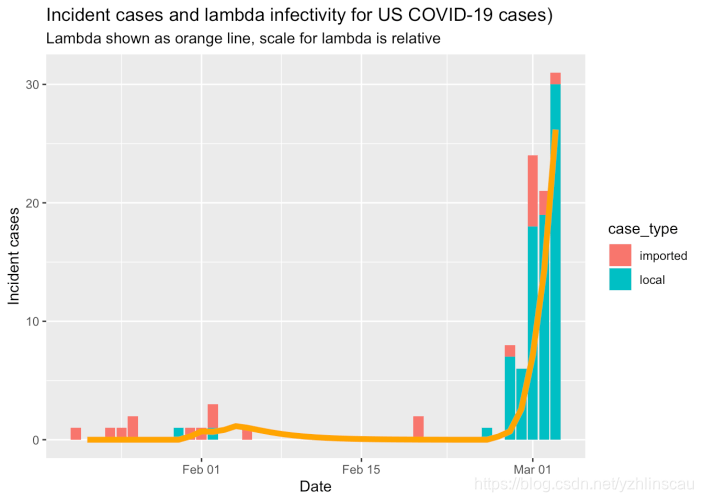
美国尚未赢得对抗COVID-19的战争,且还处于初期。
我们还可以估计再生数。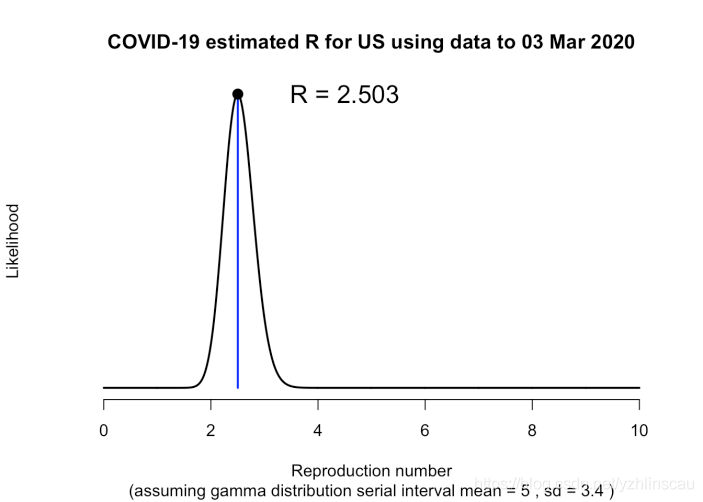
R0的估计值与WHO最近报告的估计值一致,尽管高于武汉的一些初始估计值。关键是它远高于1.0,这意味着疫情爆发正在迅速增长。
将对数线性模型拟合到流行病曲线
我们还可以使用RECON包中的函数将对数线性模型拟合到流行病曲线。通常,会拟合两个模型:一个用于增长阶段;一个用于衰减阶段。软件包中提供了使用简单和优化方法寻找流行曲线峰值的函数。 可以在这里找到示例。
就目前而言,美国的爆发仍处于增长阶段,因此我们只能拟合一条曲线。
us_incidence_fit <- incidence::fit(local_cases_obj, split = NULL)
# plot the incidence data and the model
fitplot(local_cases_obj) %>% add_incidence_fit(us_incidence_fit)
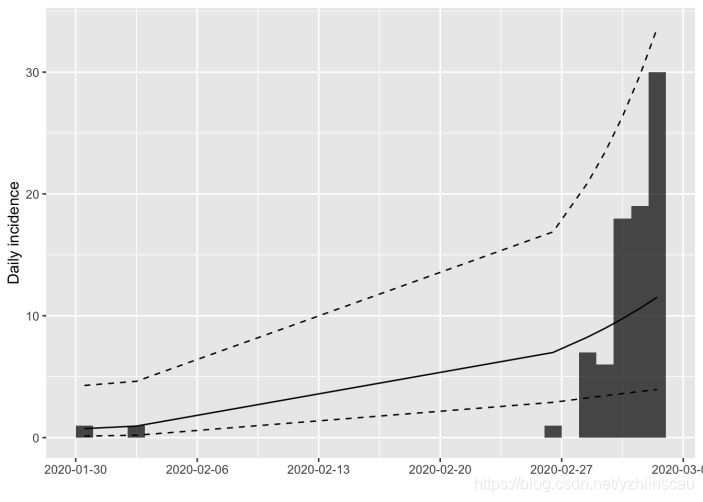
显然,这不是一个好选择,因为我们包括了少数似乎并未建立持久的本地传播链的早期病例。可以排除它们。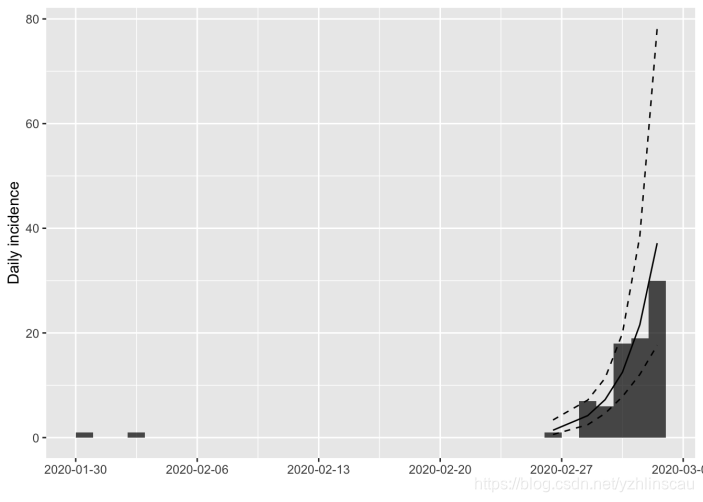
现在的拟合更好!
从该模型中,我们可以提取感兴趣的各种参数(在此初期非常初步):增长率为0.54(95%CI: 0.32 – 0.77),相当于加倍时间为1.3天(95%CI: 0.9 – 2.2天)。
这有点令人震惊,但是这些估计值几乎可以肯定是有偏差的,因为病例是根据报告日期而不是症状发作日期来制表的。我在较早的帖子中讨论了卫生当局按发病日期报告或提供数据的至关重要性。但是,随着流行病在美国的蔓延,使用报告日期引起的偏差应会减少,前提是病例的检测和报告应迅速且一致地进行。及时测试和报告病例的能力是衡量公共卫生干预能力的关键指标。
我们还可以预测下周可能会有多少病例,假设公共卫生控制未开始起作用,并且受到上述估计偏差的影响,请记住,我们的模型目前只适用于几天的数据。我们将按对数形式绘制,以便到目前为止观察到的病例不会被预测值所掩盖。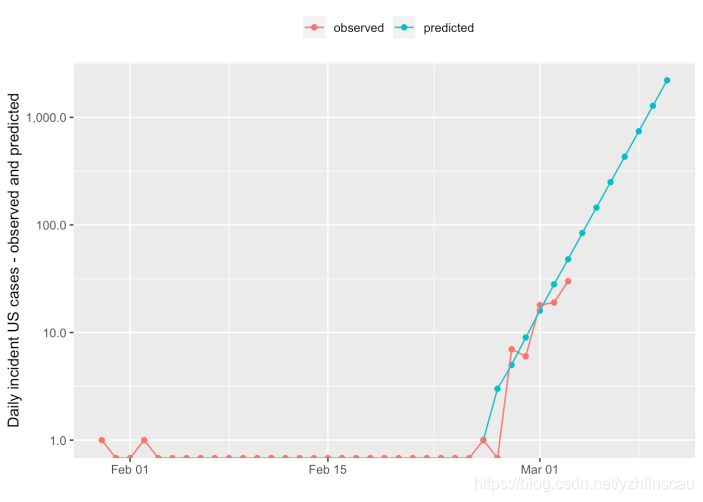
在线性范围内,如下所示: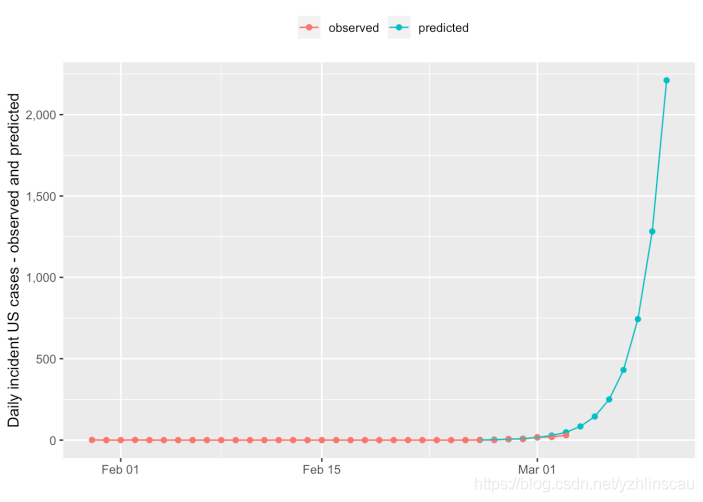
因此,根据非常粗略的初步数据,我们预测到3月10日,每天将有2211例新病例。由于之前已讨论的潜在报告日期而非开始日期的偏差,这可能高估了,但是它仍然说明了典型流行病行为的指数性质。
人们在考虑趋势时倾向于使用线性启发式方法,因此倾向于对这种指数行为感到惊讶,并且无法相应地进行规划。
估算瞬时有效再生率
EpiEstim包估计的另一个统计量是基于可调整的滑动窗口的瞬时有效再生数。 这对于评估公共卫生干预措施的效果非常有用。目前还没有足够的美国数据来估算这一点,下图是来自中国湖北省的瞬时ReRe的示例图,该图取自先前的博客文章: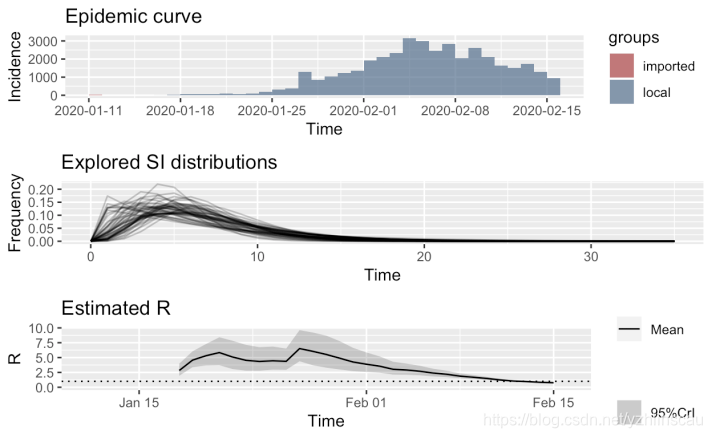
我们可以清楚地看到1月24日前后在湖北省和武汉市实施的封城措施的效果,以及在每日新发病例达到峰值之前很长一段时间内,瞬时Re开始下降的事实。这些有用性的信息可帮助政府当局保持警惕,并坚持实行不受欢迎的公共卫生措施,即使面对病例不断上升的情况。
结语
在这篇文章中,我们看到了如何使用R,tidyverse程序包,以及RECON站点提供的R包来组装COVID-19暴发数据,对其进行可视化,并从中估算一些关键统计量,这些统计量对于评估和规划针对该疾病的公共卫生措施至关重要。还有其他几个R包也可达到此目的。一小组数据科学家只需几天时间,就可以使用这些工具来构建持续的报告或决策支持工具,这些工具可以连续或至少每天更新一次,以帮助支持公共卫生当局与COVID-19的生死战。
但是我们需要立即启动,因为流行病的行为是指数级的。
https://blog.sciencenet.cn/blog-1114360-1222183.html
上一篇:利用R语言简单的时间序列模型预测韩国、日本、伊朗和意大利的疫情趋势
下一篇:基于植物试验的GBLUP模型之Echidna篇