博文
武汉新型冠状病毒2019-nCoV基因组分析
|
武汉新型冠状病毒2019-nCoV基因组分析
撰文:李杰 常熟理工学院生物与食品工程学院
责编:刘永鑫 中科院遗传发育所
写在前面
2020 年 1 月 10 日,武汉发现的新型冠状病毒的基因组被破译。该病毒测序由复旦大学生物医学研究院张永振教授领导的协作团队完成。悉尼大学的 Edward C. Holmes 代表团队上传结果,并最先在 Twitter 公布了这一消息。目前该病毒序列在 virologic.org 网站中可以查询,也已经上传到了 GenBank 中(但未公开)。序列下载地址如下:
http://virological.org/t/initial-genome-release-of-novel-coronavirus/319
研究团队同时声明:“请随时下载、共享、使用和分析此数据。如果您希望在杂志上发表使用这些数据得到的研究结果,请与我们联系。如果您还有其他疑问,请直接与我们联系。”
据1月12日世界卫生组织官微报道,中国已与世卫组织分享新型冠状病毒(2019-nCoV)全基因组序列,可以看出该新型冠状病毒目前已被命名为2019-nCoV(2019新型冠状病毒)。
上一篇文章发表后,大家关注度很高,为避免简单粗暴的以讹传讹,本次进行后续分析报道。
基因组注释
利用NCBI上的ORFfinder工具进行该病毒的基因组注释分析。

软件给出了14个主要的开放阅读框(可以简单理解为具有编码基因的能力)。下图中标明了orf1ab和四个主要结构蛋白的位置。

结构基因S的比较
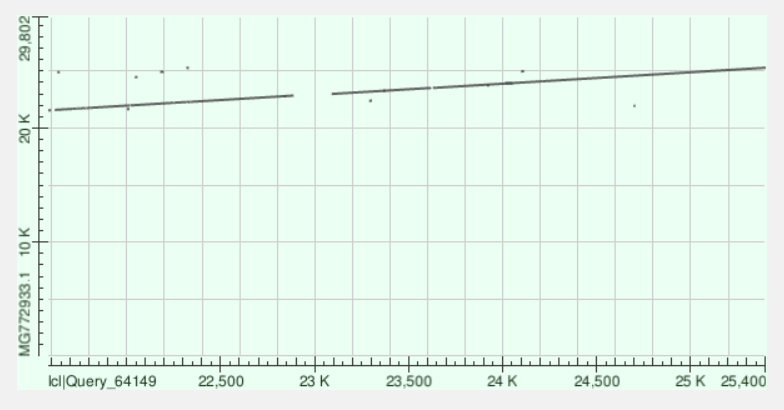
武汉毒株的S基因与bat-SL-CoVZC45的核苷酸相似性只有83.45%(这个数值应该取得是下图range1的结果,整个S基因的相似性应该低于这个数值)。
下图是核苷酸比对的情况,可以看到S基因中间有一段差异很大。
其他结构基因的比较
E基因的核苷酸序列相似性为98.68%,M基因的核苷酸序列相似性为93.42%,N基因的核苷酸序列相似性为91.03%。氨基酸序列相似性暂未分析,但结果应该跟核苷酸序列相似性相差不大。
系统进化树的构建
通过下载一系列相关的SARS-CoV、bat-SARS-CoV以及一些蝙蝠来源的α、β冠状病毒全基因组序列,通过MEGAX软件进行多重序列比对并构建了系统进化树。可以看到武汉病毒跟bat-SL-CoVZC45属于同一分支,但距离人源的SARS-CoV病毒较远。

武汉病毒与SARS-CoV的关系
画重点,武汉病毒不是SARS-CoV病毒,不是SARS-CoV病毒,不是SARS-CoV病毒,二者不能划等号。
参考文献:
1. 有关新型冠状病毒的常见问题,https://weibo.com/ttarticle/p/show?id=2309404460589488406667
2. 我国公布武汉新型冠状病毒基因序列,速度之快受到国内外广泛好评http://3g.163.com/all/article/F2N2J4N9053296CT.html
https://blog.sciencenet.cn/blog-1125809-1214288.html
上一篇:武汉新型冠状病毒是bat-SARS的变种
下一篇:不能用有漏洞的逻辑来说明2019-nCoV病毒不是人为泄露的