博文
Nature同种负密度制约论文中的统计错误
|
Nature同种负密度制约论文中的统计错误
Barro Colorado Island (BCI)这个地方,堪称群落生态学圣地。基于BCI大样地的Nature, Science大作层出不穷。今年三月份,Nature又发了一篇BCI样地中同种负密度制约效应(CNDD)和水分条件关系的论文,发现越是干旱的年份,负密度制约效应强度越弱,而湿润年份的CNDD效应越强,这是水分条件对生物多样性维持进行调控的一个案例。同种负密度制约效应,或者叫Janzen-Connell假说,无疑是生态学领域最吸引人,最有热度,最接近“物理定律”的假说之一(另一个可能就是种面积关系假说了)。不夸紧张的说,Janzen-Connell假说的存在,提升了生态学能够称之为一门科学的底气。这篇文章证明了水分对CNDD效应较强的调控作用,无疑具有重要意义,发表在Nature也是理所应当。
出于对CNDD效应的崇拜和兴趣,我细读了这篇论文,发现其结果图Fig.3b似乎有点问题。Fig.3b中,横坐标为一个幼苗的同种邻居个体的数量,纵坐标为幼苗的成活率。拟合线从黄到蓝,表示水分条件越来越好。黑色虚线表示水分条件取均值时候的拟合线。按文中所描述,Fig.3b是基于二项分布的广义线性混合模型(GLMM, 链接函数为logit)的拟合结果制图。而GLMM模型由于其期望值经过了logit转换,所以因变量Y和自变量X在原始尺度上为直线关系的可能行非常低。根据这个常识判定Fig.3b(两个坐标都是原始值)很可能有问题。而原文附件中,给出了其他图的作图代码,唯独没给Fig.3b的代码。根据图来判断,很可能是在对模型结果的反logit转换和制图中除了问题。
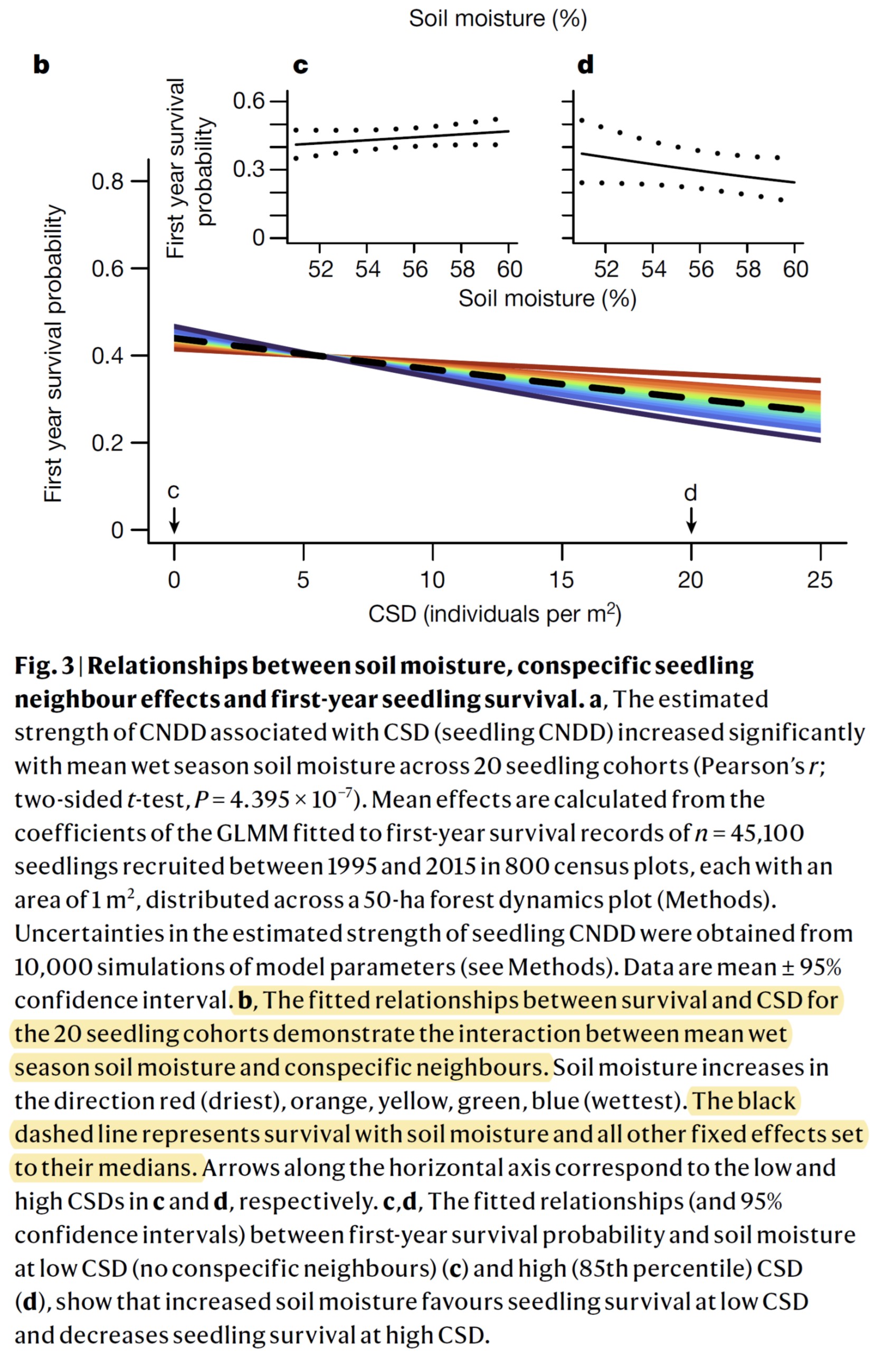
为了弄清这一问题,我重新拟合了其模型,并根据其结果,制作不同水分条件下的
存活率和同种邻居密度的关系图(代码附后)。果不其然,二者的结果图应该如下图所示。以下三种图是等价的,可以看出,跟论文中的原始图,还是有很大差别。原始尺度上(左图,从红到蓝,越来越湿润),存活率和同种邻居密度的关系不再是一条直线而是一条曲线。
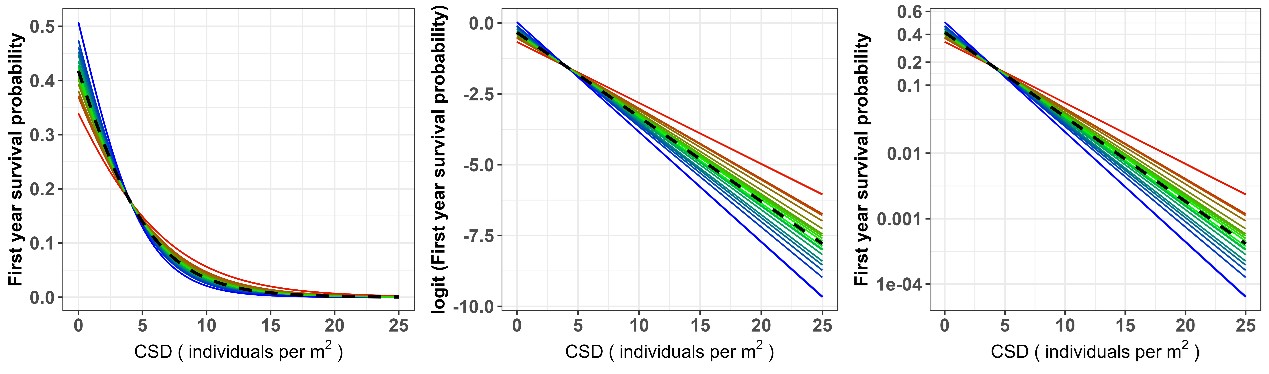
这个结果更强化了其结论,即越干旱,CNDD效应就越弱,越湿润,CNDD效应就越强,且这个作用强度远高于原文图中所示的强度。虽然结果没有根本性改变,但这个也提醒我们,纵然Nature,也会犯错,我们在使用统计模型,尤其是GLMM这类稍显复杂和前卫的模型时,一定要足够小心才行!
我前一段已经发邮件告知此文一作这个错误,没想到这哥们儿当时正在以色列,回复我说战争已经让无暇估计其他,似乎网络也没保障了。真是呜呼哀哉,但愿作者无恙,世界早日回归和平!
对这个研究或者分析方法感兴趣,想获取这个分析和作图代码的朋友可以给我发邮件(shuangzhang@rcees.ac.cn),我可以把代码发给大家,以便我们进一步交流。
参考文献:
Lebrija-Trejos, E., A. Hernández, and S. J. Wright. 2023. Effects of moisture and density-dependent interactions on tropical tree diversity. Nature 615:100-104.
最后,欢迎大家关注我的个人微信公众号:二傻统计
附代码:
## LOAD PACKAGES AND FILES ####
library(nlme)
library(lme4)
library(arm)
library(DHARMa)
library(foreach)
library(doParallel)
library(reshape2)
library(vegan)
library(cocor)
library(lubridate)
library(doBy)
Data<-read.table(file=paste("Seedl_SM.csv",sep="/"), sep=",", header=T, as.is = T)
bestmodel <- glmer(ALIVE ~ CSD + HSD.fort + CLD + HLD + SMw + CSD:SMw + HSD.fort:SMw + SpMO + SpMO:SMw
+ (1 + CSD + CLD + HLD + SMw|SPECIES)
+ (0 + HSD.fort + CSD:SMw + HSD.fort:SMw||SPECIES)
+ (1|PLOT) + (1|TRAP)
+ (1 + CSD + HSD.fort + CLD + HLD +SpMO|YEAR),
control = glmerControl(optCtrl = list(maxfun = 1e+06)),
data = Data, family = binomial, verbose = 0, nAGQ = 1)
############### fig.3 b
SMw<-sort(unique(Data$SMw))
CSD<-seq(0,25,0.1)
srv.p.sim<-list()
for (i in 1:20){
srv.p.sim[[i]]<-invlogit(fixef(bestmodel)[1] +
fixef(bestmodel)[2]*CSD+
fixef(bestmodel)[3]* SMw[i]+
fixef(bestmodel)[4]* SMw[i]*CSD
)}
mean_p<-invlogit(fixef(bestmodel)[1] +
fixef(bestmodel)[2]*CSD+
fixef(bestmodel)[3]* median(SMw)+
fixef(bestmodel)[4]* median(SMw)*CSD)
d1<-data.frame(y=srv.p.sim)
colnames(d1)<-c(1:20)
d1$CSD<-CSD
d2<-gather(d1, key="SMw", value="Pred", 1:20)
d2$moistlevel<-as.numeric(d2$SMw)
pal <- colorRampPalette(c("red","green","blue"))
my_colors <- pal(20)
########## original scale
f3b_v1<-ggplot()+
geom_line(data=d2,aes(x=CSD, y=Pred,group=SMw,color=moistlevel))+
scale_colour_gradientn(colors = my_colors)+
geom_line(aes(x=CSD,y=mean_p), size=1, color="black",
linetype = "dashed")+
theme(legend.position="none")+
ylab("First year survival probability") +
xlab(xlab)+
theme_bw()+
theme(axis.text=element_text(size=12,face="bold"),
axis.title=element_text(size=12,face="bold"))+
theme(strip.text.x = element_text(size =12),
strip.text.y = element_text(size =12),
legend.position='none')
########## logit scale
srv.p.sim2<-list()
for (i in 1:20){
srv.p.sim2[[i]]<-fixef(bestmodel)[1] +
fixef(bestmodel)[2]*CSD+
fixef(bestmodel)[3]* SMw[i]+
fixef(bestmodel)[4]* SMw[i]*CSD
}
d3<-data.frame(y=srv.p.sim2)
colnames(d3)<-c(1:20)
d3$CSD<-CSD
d4<-gather(d3, key="SMw", value="Pred_logit_scale", 1:20)
d4$moistlevel<-as.numeric(d4$SMw)
logit_mean_p<-fixef(bestmodel)[1] +
fixef(bestmodel)[2]*CSD+
fixef(bestmodel)[3]* median(SMw)+
fixef(bestmodel)[4]* median(SMw)*CSD
f3b_v2<-ggplot()+
geom_line(data=d4, aes(x=CSD, y=Pred_logit_scale,group=SMw,color=moistlevel))+
scale_colour_gradientn(colors = my_colors)+
geom_line(aes(x=CSD,y=logit_mean_p), size=1, color="black",
linetype = "dashed")+
theme(legend.position="none")+
ylab("logit (First year survival probability)") +
xlab(xlab)+
theme_bw()+
theme(axis.text=element_text(size=12,face="bold"),
axis.title=element_text(size=12,face="bold"))+
theme(strip.text.x = element_text(size =12),
strip.text.y = element_text(size =12),
legend.position='none')
########## logit scale, but relabel the y axis
f3b_v3<-ggplot()+
geom_line(data=d4, aes(x=CSD, y=Pred_logit_scale, group=SMw,color=moistlevel))+
scale_colour_gradientn(colors = my_colors)+
geom_line(aes(x=CSD,y=logit_mean_p), size=1, color="black",
linetype = "dashed")+
scale_y_continuous(breaks = c( logit(0.0001), logit(0.001), logit(0.01),logit(0.1), logit(0.2), logit(0.4), logit(0.6), logit(0.8)),
labels=c(0.0001, 0.001, 0.01, 0.1, 0.2, 0.4, 0.6, 0.8))+
theme(legend.position="none")+
ylab("First year survival probability") +
xlab(xlab)+
theme_bw()+
theme(axis.text=element_text(size=12,face="bold"),
axis.title=element_text(size=12,face="bold"))+
theme(strip.text.x = element_text(size =12),
strip.text.y = element_text(size =12),
legend.position='none')
library(cowplot)
Figure_3b_bestmodel<-plot_grid(f3b_v1, f3b_v2,f3b_v3, nrow=1, ncol=3)
ggsave("Figure_3b_bestmodel.tiff", width=11.61, height=3.42, dpi=600)
https://blog.sciencenet.cn/blog-3442043-1407938.html
上一篇:关于正态性的再答疑
下一篇:Meta 分析Science 封面双杀