博文
2021年(4):PWO-CPI:基于化合物的结构图像预测化合物与蛋白质的相互作用
||
预测化合物和蛋白质的相互作用(Compound Protein Interaction, CPI)的可以加快药物的研发与疾病的治疗。目前对于CPI的研究大多是基于序列(SMILES或FASTA)或图结构(graph)。受计算机视觉领域最新研究成果的启发,华东师范大学计算机学院张倩/钱莹等提出从一种全新的视角,即利用分子图像(image),来表征分子结构信息,从而预测CPI。相关研究成果近期在计算化学专业期刊Journal of Computational Chemistry上发表。

图1、化合物分子的表示形式。
化合物可以有多种形式表示。从图1中可以看出相较于其他分子表现形式,图像包含了更多的分子内部信息以及空间结构信息。近年来,计算机视觉领域最新研究成果证明:图像中物体的位置、几何、空间结构、相互关系等特征,可以被深度学习充分地表征,且这些特征对目标分类、检测、识别和相似目标生成等具有巨大的贡献。因此,论文在此构建了一个基于化合物分子图像特征提取的CPI模型。PWO-CPI模型图如图2所示。整个模型包含三个模块:化合物特征提取器、蛋白质特征提取器和分类器。首先构建了一个CNN的模型来学习化合物分子图像中的信息。其次在利用Word2vec模型来学习蛋白质序列的特征表示。最后通过全连接层来预测结果。
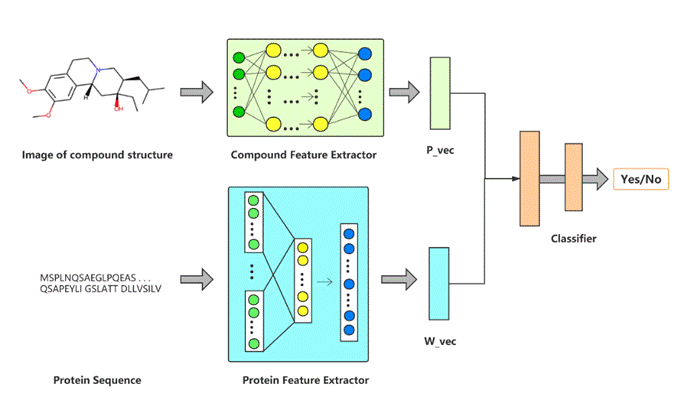
图2、PWO-CPI模型结构图。
表1. Human数据集试验结果。
| Method | AUC | Precision | Recall |
| KNN | 0.86 | 0.927 | 0.798 |
| RF | 0.94 | 0.897 | 0.861 |
| L2 | 0.911 | 0.913 | 0.867 |
| SVM | 0.91 | 0.966 | 0.969 |
| GraphDTA | 0.960±0.005 | 0.882±0.040 | 0.912±0.040 |
| GCN | 0.956±0.004 | 0.862±0.006 | 0.928±0.010 |
| GNN | 0.97 | 0.918 | 0.923 |
| TransformerCPI | 0.973±0.002 | 0.916±0.006 | 0.925±0.006 |
| PWO-CPI | 0.984±0.002 | 0.944±0.006 | 0.942±0.009 |
表2. Celegans 数据集试验结果。
| Method | AUC | Precision | Recall |
| KNN | 0.86 | 0.927 | 0.798 |
| RF | 0.94 | 0.897 | 0.861 |
| L2 | 0.911 | 0.913 | 0.867 |
| SVM | 0.91 | 0.966 | 0.969 |
| GraphDTA | 0.960±0.005 | 0.882±0.040 | 0.912±0.040 |
| GCN | 0.956±0.004 | 0.862±0.006 | 0.928±0.010 |
| GNN | 0.97 | 0.918 | 0.923 |
| TransformerCPI | 0.973±0.002 | 0.916±0.006 | 0.925±0.006 |
| PWO-CPI | 0.984±0.002 | 0.944±0.006 | 0.942±0.009 |
为了验证模型的有效性,分别在Human1和Celegans1两个数据集上进行CPI实验并取得最好的结果,试验结果如表1、表2所示。通过与传统机器学习和深度学习方法的对比,化合物结构式图像可以很好的用于表示分子。此外,为了验证分子图像表现形式的有效性,还单独将化合物提取器的模型提取出来用于DDI (Drug-Drug Interaction) 任务。通过化合物特征提取器中的CNN模型来学习两张不同药物分子图像中的信息,在Biosnap2数据集上进行试验预测药物之间是否有相互作用。根据表3中的实验结果显示,对药物分子的图像表征进行学习可以大大提高DDI的预测结果。
表3. DDI试验结果对比。
| Method | AUC | Precision | Recall |
| KNN | 0.86 | 0.927 | 0.798 |
| RF | 0.94 | 0.897 | 0.861 |
| L2 | 0.911 | 0.913 | 0.867 |
| SVM | 0.91 | 0.966 | 0.969 |
| GraphDTA | 0.960±0.005 | 0.882±0.040 | 0.912±0.040 |
| GCN | 0.956±0.004 | 0.862±0.006 | 0.928±0.010 |
| GNN | 0.97 | 0.918 | 0.923 |
| TransformerCPI | 0.973±0.002 | 0.916±0.006 | 0.925±0.006 |
| PWO-CPI | 0.984±0.002 | 0.944±0.006 | 0.942±0.009 |
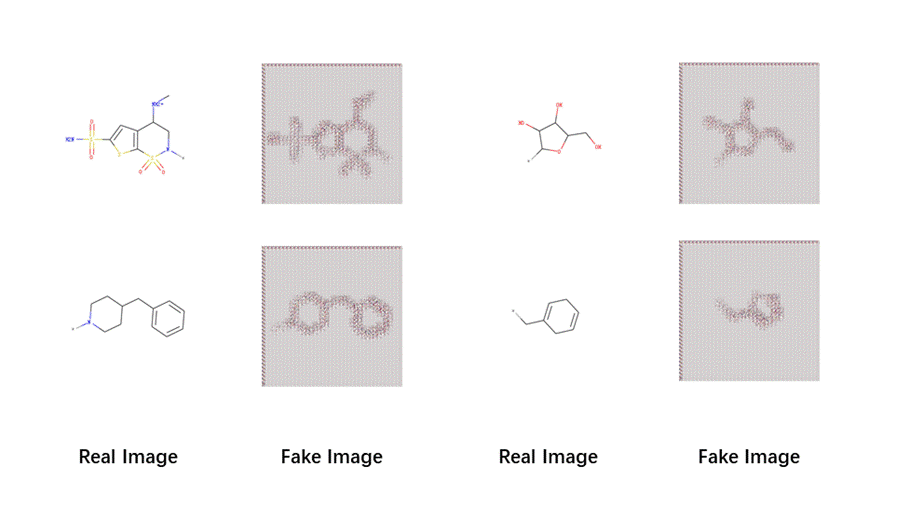
图3、通过GAN网络生成的图像和真实图像的比较。
最后构建一个对抗神经网络 (GAN) 来对分子图像进行学习,查看CNN模型是否有效学习到分子图像中的信息。通过GAN网络生成的图像和真实图像的对比如图3所示。从对比中可以看出CNN模型可以有效的学习分子的结构信息。
论文第一作者为华东师范大学计算机学院钱莹副教授,通讯作者为华东师范大学计算机学院张倩副研究员,共同作者为徐志建研究员(中国科学院上海药物研究所)、周爱民研究员(华东师范大学)、李雪莲(华东师范大学硕士)、吴坚(华东师范大学硕士研究生)。
原文链接:
http://doi.org/10.1002/jcc.26786
参考文献:
1. Liu, H.; Sun, J.; Guan, J.; Zheng, J.; Zhou, S., Improving compound–protein interaction prediction by building up highly credible negative samples. Bioinformatics 2015, 31, i221-i229.
2. Huang, K.; Xiao, C.; Hoang, T.; Glass, L.; Sun, J. Caster: Predicting drug interactions with chemical substructure representation. In Proceedings of the AAAI Conference on Artificial Intelligence, 2020; 2020; Vol. 34; pp 702-709.
https://blog.sciencenet.cn/blog-2877557-1316494.html
上一篇:2021年(3): 新冠病毒免疫逃逸机制的计算模拟及预测
下一篇:2022年(1):新冠病毒变异株Omicron刺突蛋白RBD结构域与宿主ACE2蛋白的亲和力弱于目前主流的Delta毒株